科研进展
《JIPB》发表万建民院士团队"Engineer the eukaryotic OMEGA−Fanzor systems for genome editing in plants"
发布人: 发布日期: 2025-10-10 浏览次数:
作为CRISPR系统的祖先,OMEGA同样是RNA引导 (ωRNA) 的可编程的核酸酶系统,主要包含IscB、IsrB、TnpB和Fanzor等。其中,真核生物来源的Fanzor蛋白与原核生物来源的TnpB为同源蛋白,其核酸酶特性与Cas12蛋白类似,大小仅为Cas12a蛋白的一半 (平均约620 aa),但其在植物基因组中难以实现有效的基因编辑。
近日,JIPB发表了南京农业大学万建民院士团队题为"Engineer the eukaryotic OMEGA−Fanzor systems for genome editing in plants"的研究论文 (https://doi.org/10.1111/jipb.70049)。该研究利用单个转录单元 (Single transcript unit, STU) 策略开发出适用于植物的高效OMEGA−Fanzor基因组编辑系统,并在水稻再生植株中实现了最高50.0%的编辑效率,为植物育种技术和功能基因研究提供了新工具。
本研究首先构建了基于pCAMBIA1300骨架的STU载体,与需要核酶介导sgRNA成熟的Cas9-STU系统不同,Fanzor-STU系统因其Fanzor蛋白能够加工自身mRNA以生成功能性ωRNA而省略了核酶。研究团队在水稻原生质体中测试了4种Fanzor蛋白的活性,其中NlovFz2效率为2.8%,SpuFz1和MmeFz2的效率分别为1.4%和0.9%,而GtFz1几乎未检测到活性 (图1)。随后,团队在小麦原生质体中进行了活性测试,结果显示NlovFz2和SpuFz1分别在TaDEP1靶点实现了2.9%和1.0%的编辑效率,表明了Fanzor-STU载体构建策略在不同的作物中均能适用。
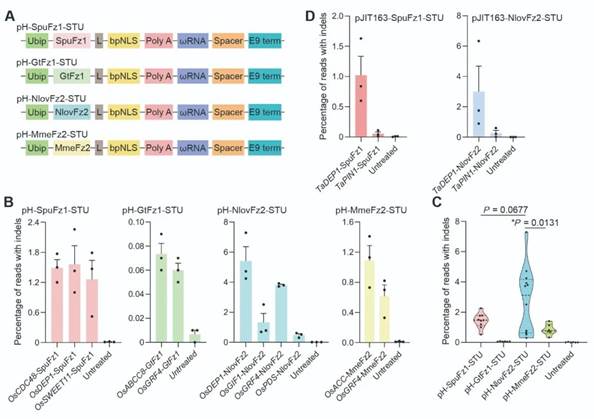
图1 OMEGA-Fanzor系统在水稻和小麦原生质体中的活性测试
此外,研究团队采用AlphaFold3对NlovFz2的结构进行预测,发现NlovFz2蛋白N端存在一个含有类NLS信号的无序区域。为了验证无序序列对编辑效率的影响,研究团队构建了四种截短类型的NlovFz2-STU载体,并通过N端带有或不含SV40 NLS来验证预测类NLS的功能是否能被替代。与对照3.3%的编辑效率相比,Nlov-Del.v1 (402 aa) 和Nlov-Del.v2 (387 aa) 的效率均有所降低,分别为0.9%和1.7%,而N端添加了SV40 NLS的Nlov-Del.v3 (403 aa) 效率也仅为1.7%;在将预测的NLS替换为SV40 NLS后,效率恢复到2.7%,且与未截断对照相比无显著差异 (P = 0.7000),而pH-NlovFz2-STUΔbpNLS在删除STU载体C端的bpNLS后,活性彻底丧失 (图2)。
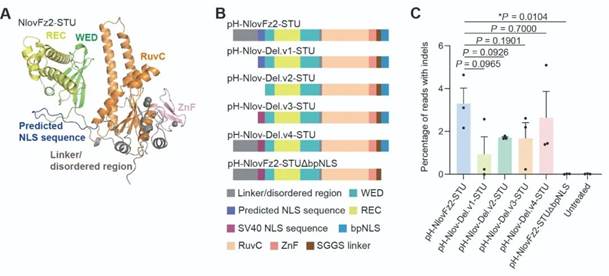
图2 NlovFz2无序区域不同截断变体对编辑效率的影响
研究团队进一步利用农杆菌介导的水稻遗传转化体系,检测了OMEGA-Fanzor系统在栽培种宁粳7号中的编辑效率。结果显示,NlovFz2的编辑效率最高 (22.5%–50.0%),随后是SpuFz1 (24.8%–34.6%) 和MmeFz2 (17.8%–18.8%),且编辑事件均为2-36 bp的DNA小片段删除。其中,NlovFz2成功在编码水稻八氢番茄红素脱氢酶OsPDS的第13外显子靶点处产生编辑,并得到了具有白化表型的突变植株 (图3)。
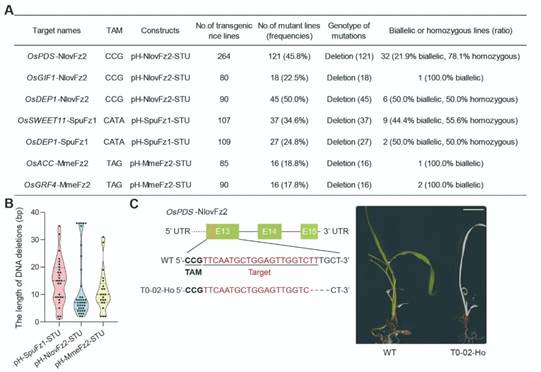
图3 OMEGA-Fanzor系统在水稻再生植株中的编辑
该研究首次在植物中系统评估并优化了真核来源的OMEGA-Fanzor系统,其中NlovFz2表现出色,具备体积小 (489 aa)、效率高、特异性强等优势。STU策略的运用使其更适合病毒介导的基因编辑 (VIGE) 和多基因编辑系统构建,未来通过ωRNA和蛋白工程进一步优化,有望丰富现有的基因编辑工具箱,为作物育种和功能基因研究提供新工具。
南京农业大学博士生纪元、孙岩和已毕业硕士生周贺杰为共同第一作者,万建民院士和李超教授为共同通讯作者。南京农业大学董小鸥教授和中国科学院遗传与发育生物学研究所王延鹏研究员等也参与了部分研究工作。该研究得到国家生物育种重大专项、国家重点研发计划、生物育种钟山实验室和南京农业大学三亚研究院引导项目等资助。

